2 注解
冠状病毒是一群具有套膜的RNA病毒,在电子显微镜下呈皇冠状,其正性单股RNA约由27000~30000多个碱基组成,为已知最大的RNA病毒,可感染人、鸡、猪、牛、鼠、猫等动物,会引发呼吸道与肠道的疾病,并引起人类轻微感冒。冠状病毒分为三大类,其中两类感染哺乳动物,另一类只感染鸟类,基本上这类病毒的宿主相当专一,主要引发呼吸道与肠道感染。2003年新发现的一种冠状病毒,为引起人类严重急性呼吸道症候群(SARS)。
严重急性呼吸综合征冠状病毒(severe acute respiratory syndrome coronavirus:SARS CoV,SCoV SARS冠状病毒)属与环状病毒(torovirus)属同为动脉炎病毒科(arteriviridae),划作套病毒(nidovims)目。冠状病毒依其抗原性差异分为3群。SARSCoV与任何一群的同源性均显不高,有的研究者将其归属于2群。Ksiazek示它可能为两群的重组病毒。2005年6月Colorado会议提议将SCoV分类为2群。
SARS冠状病毒在种属分类上属于“ssRNA positive-strand viruses”家系的“Nidovirales”族中的“Coronaviridae”系(见Taxonomy)。它是冠状病毒家族中新出现的一个子类。全长29,736bp,已知有11个编码序列(cds),而其中的一个cds(putative orf1ab polyprotein)与鼠类的肝炎病毒(murine hepatitis virus)结构类似,依据鼠类的肝炎病毒的结构模式,推断出该段cds应该编码了14个蛋白质。
3 SARS冠状病毒概述
[1][2]SARS冠状病毒(SARS-Cov),属于巢状病毒目(Order: Nidovirales),冠病毒科(Family:Coronaviridae),冠状病毒属(Genus: Coronavirus)。根据其基因组结构分类,它属于单链正义 RNA 病毒[(+)sense, ssRNA Virus]。
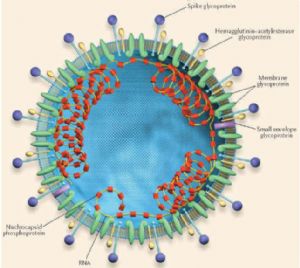
成熟的冠状病毒颗粒直径约为 60 至 220nm 不等。其形态学上最显著的特征在于,在病毒包膜(envelope)外,有明显的棒状膜外子粒('club-shaped' peplomers)。这一酷似中世纪欧洲帝王王冠(crown)的结构(如图 1),正是其”名字” - Coronavirus 的来源。
对其他已知冠状病毒的研究发现,其病毒包膜主要包括三种糖蛋白,分别命名为S 蛋白(Spike Protein)、M 蛋白(Membrane Protein)、E 蛋白(Envelope Protein)。在部分病毒株中还能找到一种 HE 蛋白(Haemagglutinin-esterase)。其中 S 蛋白即伸出包膜的棒-球形的糖蛋白,它在病毒与宿主细胞表面受体结合及介导膜融合进入细胞的过程中,起关键性作用,也是冠状病毒主要的抗原蛋白。M 蛋白则是一种跨膜蛋白,在病毒的包膜形成与出芽(budding)过程中起重要作用。E 蛋白是一种相对较小的蛋白质,主要散在分布于病毒包膜上。冠状病毒主要结构模式见图[3]。
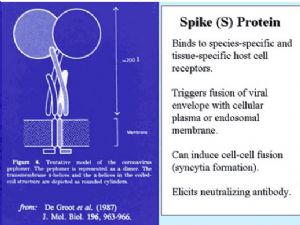 |
| S 蛋白结构模式 |
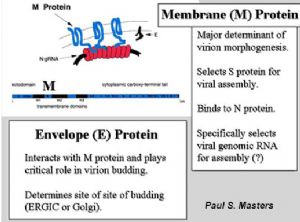 |
| M 蛋白和 E 蛋白结构模式 |
 |
| N 蛋白结构模式 |
冠状病毒入侵宿主细胞。冠状病毒的 S 蛋白是介导冠状病毒入侵宿主细胞的重要分子,
对其配体的研究亦成为关心焦点。目前认为有两类分子可能是冠状病毒的受体:氨基肽酶 N
(Aminopeptidase N)(图 3a)以及鼠类癌胚抗原细胞黏附分子 1(murine CEACAM1)(图
3b)。它们通过与冠状病毒 S 蛋白结合,诱导冠状病毒 S 蛋白发生结构上的变化,暴露穿膜
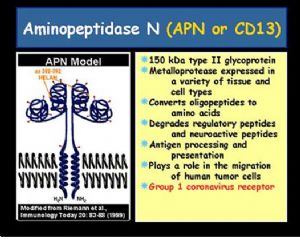 |
| 3a.氨基肽酶 N |
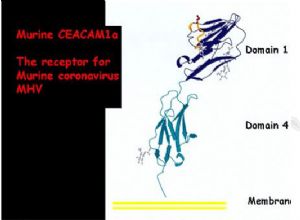 |
| 3b. murine CEACAM1 |
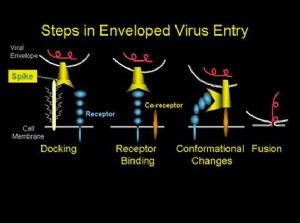 |
| 3c.入侵过程 |
位于病毒颗粒中央的不规则核酸部分即病毒基因组,其上结合有核壳体蛋白(N蛋白,Nucleocapsid Protein)。冠状病毒基因组 RNA(Genomic RNA)是一个无分段的,正义单链 RNA,长度一般在 27-31kb。该 RNA 链具有一个正链 RNA 病毒特有的重要结构特征:即 RNA 链 5`端有甲基化帽(methylated cap)、3`端有 PolyA 尾的结构。这一结构,和真核mRNA 非常相近,也是其基因组 RNA 自身即可发挥翻译模板作用的重要结构基础。冠状病毒进入细胞后的转录、翻译、复制、装配与出芽过程,如图 4 所示。值得特别指出的是:冠状病毒成熟粒子中,并不存在RNA病毒复制所需的 RNA 聚合酶(Viral RNA polymerase)。因此,它进入宿主细胞之后,首先将直接以病毒基因组 RNA 为翻译模板,表达出病毒 RNA 聚合酶。然后才利用该酶完成负链亚基因组 RNA (sub-genomic RNA) 的转录合成、各结构蛋白 mRNA 的合成,以及病毒基因组 RNA 的复制。另一个需要说明的特点是:冠状病毒各个结构蛋白成熟的 mRNA 合成,并不存在转录后的修饰剪切过程,而是直接在初次转录过程中,通过 RNA 聚合酶和一些转录因子,以一种“不连续转录”(discontinuous transcription)的机制,通过识别特定的转录调控序列(transcriptionregulating sequences ,TSR),有选择性地从负义链 RNA 上,一次性转录得到构成一个成熟 mRNA 的全部组成部分。
结构蛋白和基因组 RNA 复制完成后,将在宿主细胞内质网处装配(assembly)生成新的冠状病毒颗粒,并通过高尔基体分泌至细胞外,完成其生命周期。
关于导致 SARS 的病原体,在其研究之初,全球各地的研究组曾经存在过多种推断。3月22日,香港大学微生物系首先利用非洲绿喉肾脏细胞(African Green Monkey KidneyCells, Vero E6),从一个感染者的肺组织中分离到了一种未知的病毒[4],推测其很可能即为致病原。WHO 的研究网络随即集中对该病毒进行了分析,并且在3月27日的简报中首次提到了”病原体可能是冠状病毒的一种”[5]。此后两周内,该研究网络的多个实验室对SARS进行了临床标本、病理组织与影像学、病毒分离培养、血清学与免疫学诊断、分子生物学与遗传同源性等多方面的深入研究与鉴定[6][7][8][9],最终确认:一种新的冠状病毒(Coronavirus),
4 SARS 冠状病毒基因组学研究
[10][11][12]4月12日,加拿大 British Columbia Cancer Agency’s Genome Sciences Centre 首先完成了SARS 冠状病毒的全基因组测序(Accession Number: AY274119)。我国中国科学院华大基因组研究中心,也于4月16日完成了5个SARS病毒分离株的测序工作。截至5月9日,已提交到 GeneBank 的完整 SARS 基因组序列达11条。其基本信息见表 1。NCBI 参照上述各 序 列 , 以Tor2基因组序列为蓝本 , 修正给出了SARS的基因组参考序列(AC:NC_004718)。
| 基因组序列名称 | AC 注册号 | 序列长度 | 测序国家/地区 | 最后修改日期 |
| TOR2 | AY274119 | 29751 bp | Canada | 2003.4.30 |
| Urbani | AY278741 | 29727 bp | U.S. | 2003.4.21 |
| BJ01 | AY278488 | 29725 bp | China | 2003.5.1 |
| HKU-39849 | AY278491 | 29742 bp | HongKong | 2003.4.18 |
| CUHK-W1 | AY278554 | 29736 bp | HongKong | 2003.4.28 |
| CUHK-Su10 | AY282752 | 29736 bp | HongKong | 2003.5.7 |
| isolate SIN2500 | AY283794 | 29711 bp | Singapore | 2003.5.9 |
| isolate SIN2677 | AY283795 | 29705 bp | Singapore | 2003.5.9 |
| isolate SIN2679 | AY283796 | 29711 bp | Singapore | 2003.5.9 |
| isolate SIN2748 | AY283797 | 29705 bp | Singapore | 2003.5.9 |
| isolate SIN2774 | AY283798 | 29711 bp | Singapore | 2003.5.9 |
表1.已完成测序的 SARS 冠状病毒全基因组序列 (截至 2003.5.9)
通过与已知冠状病毒基因组的比较分析,三个完成测序的研究组-USCCDC、加拿大BCCA 基因组研究所、北京华大基因组研究中心,分别基于 Urbani/Tor2/BJ01的序列,对 SARS冠状病毒的基因组结构进行了分析预测,并发表了各自的工作。三个病毒株的分析结果基本一致,认为 SARS 冠状病毒基因组属于典型的缺乏 HE 蛋白的冠状病毒[HE(-)-Coronavirus]
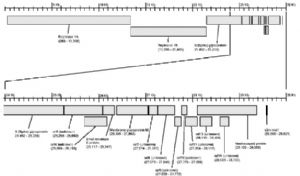
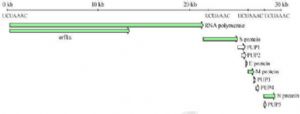
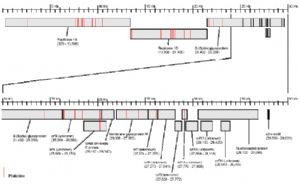
基因组结构。其基因组 5’端约三分之二的区域,编码病毒 RNA 聚合酶复合蛋白;后三分之一的区域,编码病毒结构蛋白,按基因组上的排列顺序依次为 S 蛋白、E 蛋白、M 蛋白、N蛋白;未发现 HE 蛋白编码序列。已发表的 USCDC、加拿大、北京华大的基因组结构图分别如图 5-7 所示。
上述三篇基因组分析论文10-12均指出在结构蛋白编码区可能的开放读码框(Open Reading Frame, ORF)中,存在已有蛋白质序列数据库中未找到任何同源序列的未知蛋白(Predicted Unknown Protein, PUP)。但是在具体的PUP数目和位置上,各组之间的结论存在一定的差异。其中USCDC对Urbani和北京华大对BJ01的分析,均报道发现了5个PUP;加拿大Tor2的分析则报道发现了9个可能的未知ORF和1个s2m motif。本研究在4月26日,即基于Genebank已有的基因组序列,参考 NCBI 的简要注释,对其进行了系统的同源性检索分析,并在第一时间发布,分析其结果与随后发表的上述三篇论文基本一致20。现以加拿大Tor2的分析结果为参照,将三地对于未知蛋白的结果罗列如下:
Predicted Unknown ORF | Start-End | AA length | Urbani Corresponding | BJ01 Corresponding |
| Orf3 | 25,268–26,092 | 274 | X1 | PUP1 |
| Orf4 | 25,689–26,153 | 154 | X2 | PUP2 |
| Orf7 | 27,074–27,265 | 63 | X3 | PUP3 |
| Orf8 | 27,273–27,641 | 122 | X4 | PUP4 |
| Orf9 | 27,638–27,772 | 44 | N/A | N/A |
| Orf10 | 27,779–27,898 | 39 | N/A | N/A |
| Orf11 | 27,864–28,118 | 84 | X5 | N/A |
| Orf13 | 28,130–28,426 | 98 | N/A | PUP5 |
| Orf14 | 28,583–28,795 | 70 | N/A | N/A |
表2. Tor2 / Urbani /BJ01 基因组中未知蛋白 ORF 的预测对比
此外,加拿大和美国的研究组,均对冠状病毒基因组的5’端非翻译区(UTR)和各个 ORF起始密码子上游的区域进行了分析,以期待寻找到类似于其他冠状病毒”UCUAAAC”的,参与“不连续转录”识别的特征性TRS(转录调控序列)。两个研究组均在orf1a、S蛋白、X1(即 Tor2 ORF3 )、M 蛋白、X4(即 Tor2 ORF8)、X5(即 Tor2 ORF9)和 N 蛋白上游,发现了一个保守的8个核苷酸的 TRS――”AAACGAAC”。另外,加拿大的研究还显示在其他几个ORF上游,也有类似的TRS。(如表 3 所示)。上述发现,进一步支持了冠状病毒的不连续转录模型,也为ORF预测的准确性提供了强有力的证据。
另外,上述诸研究组的分析工作还发现了一些细节的结构现象。加拿大的研究提到在 3’端非编码区,存在一个保守的 s2m motif。这个在某些病毒中广泛存在的结构可能对于了解SARS 冠状病毒的来源有提示作用[13]。中国 BJ01 的分析结果还发现了至少四个单位长度在 7个碱基以上的回文结构(palindromes)、140 多个可能形成发夹结构(hairpin)的片段,以及发现 BJ01 基因组上 155-211/861-920 这两个区域内的大约 60 个碱基出现重复现象12。上述有趣的基因组现象的生物学意义均有待进一步研究证实。
5 SARS CoV 突变与 SARS 系统发生关系(Phylogenetic)分析
由于 SARS 是一种突发的严重传染性疾病,短期内在多个国家均有感染及死亡报道。因此,分析比较不同地区 SARS 病毒的序列差异与突变状况,对于了解疾病的传染过程、病毒的毒力变化以及控制该疾病的传播,都具有非常重要的意义。另外,运用比较基因组学的方法,了解 SARS 冠状病毒与已知冠状病毒的系统发生关系,以确定 SARS Coronavirus 的可能来源与种属分类,也是 SARS 研究的重要课题。
本研究组于 4 月底,利用当时已发表的 9 个不同来源的 SARS 基因组序列,对其 S 蛋白的编码区序列多样性进行了分析,并以 S 蛋白质编码基因为对象,绘制出了序列已公布的几个 SARS 病毒株的系统发生树[14]。5 月 1 日,在北京基因组中心提交 BJ01 全基因组序列之后,随即根据新的 5 条全基因组序列修正了上述 S 蛋白基因组序列突变分析,并进行了全基因组的碱基替换与氨基酸突变分析[15][16]。所得结果,与北京基因组中心随后发表的论文所述基本一致(碱基替换分布如图 8)。
北京基因组中心在其发表的工作中,着重比较分析了 BJ01/Urbani/Tor2/CUHK/HKU 等五个病毒株全基因组的碱基替换(substitution)情况及其对编码氨基酸的影响。总共发现了31个碱基替换位点, 计算总突变率约为.0.10%; 其中有义突变(non-synonymous substitution)15个[17]。具体突变的数目及分布情况见表 4。
| 替换位置(ORF) | 发生替换区域长度(nt) | 替换数目 | 替换率(%) |
| RNA polymerase | 21220 | 19 | 0.09 |
| S protein | 3767 | 4 | 0.11 |
| M protein | 665 | 2 | 0.30 |
| PUP1 | 824 | 4 | 0.49 |
| PUP3 | 191 | 1 | 0.52 |
| Non-ORF | 1 | ||
| 合计 | 29725 | 31 | 0.10 |
表 4. BJ01/Urbani/Tor2/CUHK/HKU 全基因组碱基替换分析明细表
整体突变率显示,不同地源的SARS冠状病毒基因组,至少在上述病毒株采样期间,保持了相对稳定。从绝对数量上看,突变部位主要集中在RNA聚合酶区域,但是考虑到突变ORF的长度,则显示结构蛋白编码区的突变率要大大高于聚合酶编码区,未知蛋白质区域的突变率高于结构蛋白。这一结构上的突变分布规律,与病毒功能上的稳定性具有一定的一致性。
上述突变规律的研究,对于SARS冠状病毒的防治有着十分重要的意义:首先,RNA聚合酶的保守性,进一步证实了病毒 RNA 聚合酶合成的独立性,也提示RNA聚合酶可能为抗SARS药物设计提供重要靶点。抗HIV药物中的聚合酶抑制剂的成功思路,可能也适用于SARS冠状病毒。其次,S蛋白质的突变,尤其是高比例的有义突变(3/4),提示S蛋白可能是病毒诱发机体免疫系统生成抗体,及发生细胞毒性的T细胞免疫反应的主要抗原蛋白。这对于了解SARS的急性损伤机制,制备SARS冠状病毒特异性抗体及疫苗,都有着十分重要的意义[17]。
另外,新加坡基因组中心于5月9日发表了对14条SARS全长或部分基因组序列的比较分析结果,对世界各地 SARS病毒的突变及演化关系进行了深入分析[18]。虽然由于其选用的BJ01的序列为5月 1 日修正之前的数据,使得其突变分析结果有待改进,但是他们对于各地SARS病毒相互之间演化关系的推断,还是具有相当重要的启发意义和较高参考价值的。
从上述分析工作来看,SARS 冠状病毒是一个比较稳定的病毒(但也不排除在未来其会有大规模的变异)。这个结果为我们提供了喜忧参半的信息:其稳定性有利于机体针对病毒特异性抗体的生成,降低二次感染可能性,并增加了疫苗研究的可行性;但同时也意味着病毒随传代毒力减低的可能性减小,给疾病的流行控制增加了难度。当然,要回答“SARS 病毒变还是不变?怎么变?”的问题,仅仅这些简单而有限的分析是远远不够的,还需要依赖于对不同地域、不同时间、不同感染者来源的更多病毒株的测序资料,以及更为广泛深入的临床与实验研究。
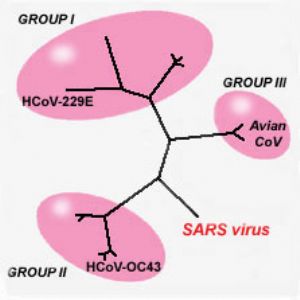
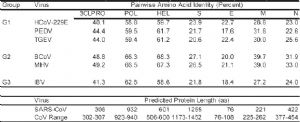
在系统发生(Phylogenetic)方面,已知的冠状病毒,根据血清学证据和种属发生学规律,分为 3 个 Group。其中 Group1,2 包括有多种哺乳动物冠状病毒,Group3 仅有鸟感染性支气管炎病毒(Avian infectious bronchitis virus, IBV)。序列同源性的分析表明,SARS 冠状病毒与已知各种冠状病毒的同源性都不高[19](见表 5)。
因此,作为一种新发现的冠状病毒,确定SARS CoV的系统发生学位置与种属关系,是病毒学研究的重要课题。在缺乏血清学系统认证的情况下,前面提及的美、加、中三个研究组在发表各自基因组分析结果的同时,都基于核酸或冠状病毒重要编码蛋白的氨基酸序列,对SARS CoV进行了系统发生学分析(Phylogenetic Analyses)。北京研究组的系统发生树,是基于Genebank中已有收载的17种不同的冠状病毒 RNA 聚合酶的核酸和氨基酸序列;USCDC则利用全基因组序列已知的 7 种冠状病毒的6个重要蛋白质氨基酸序列,绘制系统发生树(见图 9),加拿大的工作与USCDC基本类似[19]。上述工作均显示SARS CoV无法归于现有的任何一个Group,且在系统发生上与其他3个 Group 的成员基本上是等距离的。这一结果与序列同源比对的数据也是一致的。北京大学疾病基因研究中心生物信息组,在4月底也曾基于SARS CoV的S蛋白和orf1a序列,对其进行了同源比对和种系进化分析[20],其结论得到了随后发表论文的证实。
6 最新科学文献
- SARS综述:SARS病毒基因组与侵入宿主细胞研究近况
- SARS综述:SARS非典型肺炎的临床和治疗进展
- The Preliminary Analysis of SARS Co-V-Host Cell Interaction(CMBI-PUCHGSince 5.7)
- The Preliminary Analysis of SARS Coronavirus Genome and Proteins(CMBI-PUCHGSince 4.26)
- Interpretation of diagnostic laboratory tests for severe acute respiratory syndrome: the Toronto experience(CMAJ-2004.1.6)
- Laboratory tests for SARS: Powerful or peripheral?(CMAJ-2004.1.6)
- Severe acute respiratory syndrome (SARS)—paradigm of an emerging viral infection(J Clin Virology-2004,vol29)
- Biosynthesis, Purification, and Substrate Specificity of Severe Acute Respiratory Syndrome Coronavirus 3C-like Proteinase(JBC-2004.1)
- Evaluation of Control Measures Implemented in the Severe Acute Respiratory Syndrome Outbreak in Beijing,2003(JAMA-12.24)
- Interferon Alfacon-1 plus Corticosteroids in Severe Acute Respiratory Syndrome(JAMA-12.24)
- Early diagnosis of SARS Coronavirus infection by real time RT-PCR(J Clin Virology-2003,vol28)
- Severe Acute Respiratory Syndrome (SARS)-Editorial(J Clin Virology-2003,vol28)
- SARS-coronavirus replicates in mononuclear cells of peripheral blood (PBMCs) from SARS patients(J Clin Virology-2003,vol28)
- SARS virus infection of cats and ferrets(Nature-10.30)
- Reverse genetics with a full-length infectious cDNA of severe acute respiratory syndrome coronavirus(PNAS-10.28)
- Isolation and Characterization of Viruses Related to the SARS Coronavirus from Animals in Southern China(Science-10.10)
- Isolation and Characterization of Viruses Related to the SARS Coronavirus from Animals in Southern China(Sciencexpress-9.4)
- Possible role of an animal vector in the SARS outbreak at Amoy Gardens(Lancet-8.16)
- Managing SARS(N Engl J Med-8.14)
- SARS in Hong Kong(N Engl J Med-8.14)
- SARS and the Internet(N Engl J Med-8.14)
- A surfeit of SARS(Lancet-8.12)
- SARS: Epidemiology, Clinical Presentation, Management, and Infection Control Measures(Mayo Clin Proc-7)
- SARS: 1918 Revisited? The Urgent Need for Global Collaboration in Public Health(Mayo Clin Proc-7)
- Fatal Aspergillosis in a Patient with SARS Who Was Treated with Corticosteroids(N Engl J Med-7.31)
- Profile of Specific Antibodies to the SARS-Associated Coronavirus(N Engl J Med-7.31)
- Newly discovered coronavirus as the primary cause of severe acute respiratory syndrome(Lancet-7.26)
- Treatment of SARS with human interferons(Lancet-7.26)
- SARS — Looking Back over the First 100 Days(N Engl J Med-7.24)
- SARS, What have we learned?(Nature-7.10)
- Coronavirus Genomic-Sequence Variations and the Epidemiology of the Severe Acute Respiratory Syndrome(N Engl J Med-7.10)
8 参考资料
- ^ [1] ."Department of Microbiology, University of Leicester".
- ^ [2] Ehud Lavi, Susan R.Weiss, Susan T."The Nidoviruses (Coronaviruses and Arteriviruses)".Hingley, ADVANCES IN EXPERIMENTAL MEDICINE AND BIOLOGY, 494 October 2001 Hardbound 742 pp.ISBN 0-306-46634-1
- ^ [3] Holmes KV."SARS-associated coronavirus".N Engl J Med 2003 May 15;348(20):1948-51
- ^ [4] WHO."SARS virus isolated, new diagnostic test producing reliable results".March 22,
- ^ [5] WHO."SARS virus close to conclusive identification, new tests for rapid diagnosis ready soon".March 27, 2003
- ^ [6] J S M Peiris, S T Lai, L L M Poon etc."Coronavirus as a possible cause of severe acute".Lancet, Published online April 8, 2003
- ^ [7] Christian Drosten, Stephan Günther, Wolfgang Preiser etc."Identification of a Novel".NEJM, published at www.nejm.org on April 10, 2003.
- ^ [8] Thomas G.Ksiazek, Dean Erdman, Cynthia Goldsmith etc."A Novel Coronavirus Associated".NEJM, published at www.nejm.org on April 10,2003
- ^ [9] Susan M.Poutanen, Donald E.Low, Bonnie Henry etc."Identification of Severe Acute".NEJM, published at www.nejm.org on March 31, 2003
- ^ [10] ." Paul A.Rota,M.Steven Oberste, Stephan S.Monroe etc."Characterization of a Novel Coronavirus Associated with Severe Acute Respiratory Syndrome".Science, www.sciencexpress.org, 1 May 2003".
- ^ [11] Marco A.Marra, Steven J.M.Jones,Caroline R.Astell etc."The Genome Sequence of the".Science, www.sciencexpress.org 1 May 2003
- ^ [12] QIN E’de, ZHU Qingyu, WANG Jian, LI Wei etc."A complete sequence and comparative".Chinese Science Bulletin 2003 Vol.48 No.10 941-948
- ^ [13] Marco A.Marra, Steven J.M.Jones,Caroline R.Astell etc."The Genome Sequence of the SARS-Associated Coronavirus".Science, www.sciencexpress.org 1 May 2003
- ^ [14] Qipeng Zhang, Wei Rui, Lei Shi,Ming Lu."The Preliminary Analysis of the Mutation of SARS Coronaviruses Genes' Sequences".CMBI,May 1, 2003
- ^ [15] Qipeng Zhang, Wei Rui, Lei Shi,Ming Lu."The Attachment of Analysis of the Mutation of SARS Coronaviruses Genes'Sequences".CMBI,May 4, 2003
- ^ [16] Qipeng Zhang, Lei Shi, Wei Rui, Ming Lu.."The Preliminary analysis of SARS associated Coronavirus Complete Genome's Mutation".CMBI,May 4, 2003
- ^ [17] QIN E’de, ZHU Qingyu, WANG Jian, LI Wei etc."A complete sequence and comparative analysis of a SARS-associated virus (Isolate BJ01)".Chinese Science Bulletin 2003 Vol.48 No.10 941-948
- ^ [18] YiJun Ruan, Chia Lin Wei, Ling Ai Ee etc."Comparative full-length genome sequence analysis of 14 SARS coronavirus isolates and common mutations associated with putative origins of infection".Lancet, Published online May 9, 2003
- ^ [19] Paul A.Rota,M.Steven Oberste, Stephan S.Monroe etc."Characterization of a Novel Coronavirus Associated with Severe Acute Respiratory Syndrome".Science, www.sciencexpress.org, 1 May 2003
- ^ [20] ."SARS 冠状病毒预测蛋白的种系进化分析".北京大学疾病基因研究中心 April 30, 2003